Xin phép anh/chị hôm nay chúng ta đàm luận về DNA sequencing – tạm dịch là “giải trình tự DNA”.
Tế bào của tất cả các loài sinh vật trên Trái Đất chứa trong lõi của nó một bộ mã – bộ mã này chính là DNA. Thế bộ mã này có tác dụng gì? Tế bào “đọc” mã DNA để “sản xuất” ra protein. Và tập hợp các protein chịu trách nhiệm “điều hành” sự sống. Đấy, đơn giản là vậy 😊.
~
Để giúp anh/chị quyết định có đọc tiếp hay không, tôi xin phép cung cấp các thông tin liên quan đến bài post này như sau:
- Chủ đề: Bioinformatics, DNA (DeoxyriboNucleic Acid)
- Tính thời sự: tháng 4/2022
- Thời gian đọc: 6 phút, không tính thời gian uống cà phê
🧬
⓪ Đề dẫn.
Các nhà bác học đã phát hiện ra mô hình cấu trúc DNA từ năm 1953 (tham khảo). Theo mô hình này, DNA được cấu tạo bởi hai chuỗi nucleotide xoắn kép (double-helix), liên kết với nhau bằng liên kết hydro và chạy ngược chiều nhau. Mỗi sợi được cấu tạo tổ hợp từ bốn nucleotide cơ sở (base): adenine (A), cytosine (C), guanine (G) và thymine (T).
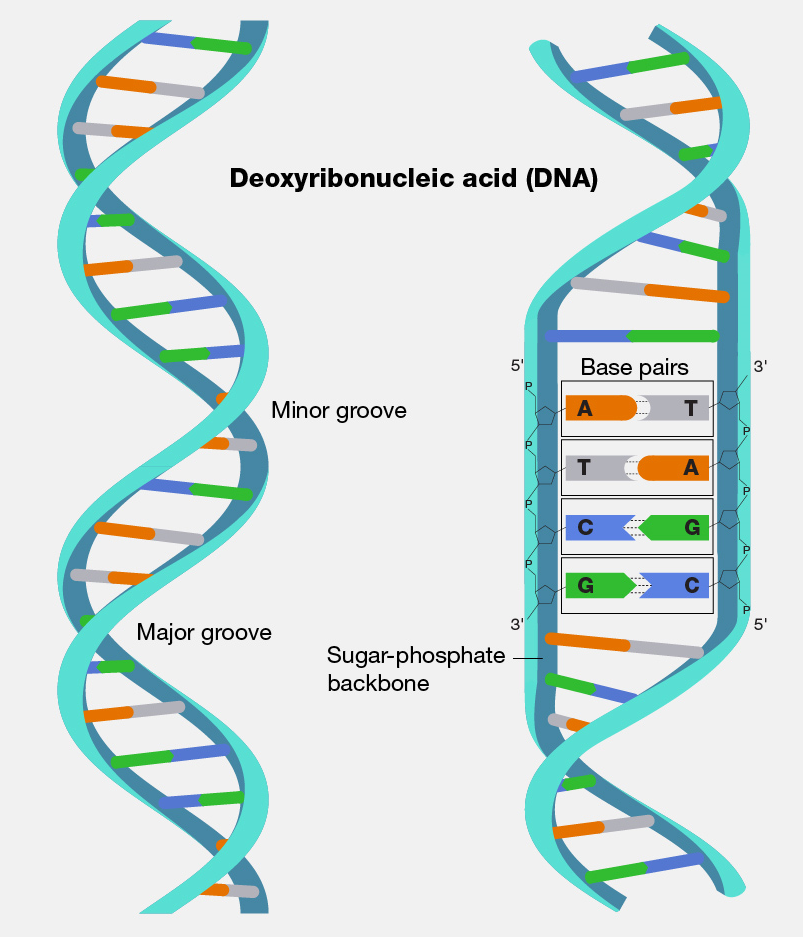
Minh họa cấu trúc xoắn kép (double helix) của DNA. Nguồn.
-
Dưới góc độ ngôn ngữ máy tính thì mỗi một sợi được tạo thành từ chuỗi các cơ số (base) mà mỗi một cơ số được chọn từ tập hợp các mã {A, C, G, T}. Như vậy, một sợi DNA là một chuỗi các ký tự mà mỗi một ký tự thuộc tập hợp {A, C, G, T}. Điểm đặc biệt là hai sợi xoắn kép này không độc lập mà bổ sung cho nhau theo cấu trúc redundant (dư thừa). Nếu trên một sợi có cơ số (base) A thì trên sợi kia tương ứng sẽ là cơ số (base) T (và ngược lại). Nếu trên một sợi có cơ số (base) C thì sợi kia tương ứng sẽ là cơ số (base) G (và ngược lại).
A ↔ T
C ↔ G
Các cơ số được “đọc” theo hai chiều ngược nhau. Trong hình minh họa ở trên, chúng ta viết chuỗi sợi kép như sau (chúng ta đọc các cơ số từ đầu 5’ đến đầu 3’ – xem hình vẽ minh họa ở trên):
<----
CGAT
||||
ATCG
---->
-
Để thống nhất về mặt ngôn từ, trong ví dụ trên, mẫu DNA này có 4 base pair (cặp cơ số) – viết tắt là bp. Để đơn giản hóa, thường người ta chỉ viết trình tự của một sợi (trình tự của sợi còn lại được suy ra từ sợi kia).
-
Mô hình cấu trúc DNA đã được biết - nhưng làm thế nào để biết được trình tự gốc các bp nằm trong “chuỗi” lại là một câu chuyện khác. Như vậy, vấn đề của DNA sequencing là xác định được trình tự DNA.
🧬
① Chiều dài gen?
⁇ Cái tiếp theo gây tò mò cho chúng ta là các loài sinh vật, bộ gen (DNA) của chúng có chiều dài là bao nhiêu, tính theo bp? (Nguồn)
- Gen người: khoảng 3 tỷ bp.
- Chim: trên dưới 1 tỷ bp.
- Vi khuẩn (bacteria): khoảng 3 triệu bp.
- Bò sát: dao động từ 2-4 tỷ bp
- Động vật có vú (mammals): 3.5 tỷ bp.
- Động vật lưỡng cư (amphibian: ếch, cóc, sa giông và kỳ nhông): 32 tỷ bp (gấp 10 lần gen người!).
- Loài hoa paris japonica: 149 tỷ bp (gấp 50 lần gen người!).
- …
Thế các loài virus có bao nhiêu bp trong gen (RNA) của chúng? Một vài ví dụ:
- Sars-cov-2 (virus gây ra đại dịch Covid-19): 29.903 bp (nguồn).
- Hepatitis B (virus gây viêm gan B): khoảng 3.200 bp (nguồn).
- Ebola (virus gây bệnh Ebola): khoảng 19,000 bp (nguồn).
Như vậy, có thể thấy là độ dài của DNA biến động từ hàng ngàn đến hàng trăm tỷ bp.
🧬
② Độ phức tạp của DNA sequencing
⁇ Câu hỏi: Giải trình tự DNA có phức tạp không? Để trả lời câu hỏi này, tốt nhất chúng ta lấy ví dụ từ thực tế: Human Genome Project – Dự án bản đồ gen người (nguồn):
- Năm 1990 Quốc hội Mỹ phê duyệt dự án Human Genome Project, dự kiến kinh phí 3 tỷ USD (tính theo tỷ giá năm 1991) trong thời gian 15 năm (1990-2005).
- Dự án hoàn thành năm 2003, trước thời hạn 2 năm, với kinh phí là 2.7 tỷ USD. (Nếu tính cả lạm phát, người ta ước tính là chi phí này tương đương 5 tỷ USD vào năm 2018.)
- Có 20 tổ chức, cơ sở nghiên cứu tham gia dự án từ các quốc gia là Hoa Kỳ (12), Đức (3), Nhật (2), Anh (1), Pháp (1), Trung Quốc (1).
- Tuy được gọi là hoàn thành vào năm 2003 nhưng vào thời điểm đó người ta mới chỉ giải được trình tự của 92.1% bộ gen. Các đoạn mã mà người ta chưa giải được trình tự được gọi là gap (lỗ hổng).
- Đến tháng 3/2009 vẫn còn 300 gaps, năm 2015 vẫn còn 160, tháng 5/2020 còn 79, năm 2021 còn 5 và vào tháng 4/2022 thì người ta đã hoàn tất bản đồ gen người.
- Như vậy, thời gian thực tế kéo dài đến 32 năm, mặc dù kể từ năm 2003, người ta không tập trung nhiều nguồn lực như trước.
- Để cảm nhận được tầm quan trọng của việc giải mã DNA (giải mã sự sống) mời anh/chị tham khảo video thông báo của Nhà trắng (ngày 26/6/2000) về bản thảo Bản đồ gen người với sự tham gia của Bill Clinton (Tổng thống Hoa Kỳ), Tony Blair (Thủ tướng Anh), nhà khoa học Francis Collins (lúc đó là Giám đốc National Human Genome Research Institute), Craig Venter (được biết đến là người đầu tiên đưa ra bản thảo Bản đồ gen người). Người ta xem đây là một bước đột phá của nhân loại.
Trả lời: Giải trình tự DNA rất phức tạp 😊!
🧬
③ Nguyên lý hoạt động của DNA sequencing
⁇ Thứ tò mò tiếp theo là cách thức hoạt động của máy giải trình tự DNA (DNA Sequencer)? Chúng ta có thể hình dung một cách đơn giản rằng khi cho đầu vào là một mẫu DNA thì máy Sequencer sẽ cho đầu ra là một chuỗi các ký tự tổ hợp từ {A, C, G, T}.
Mẫu DNA → [Sequencer] → Trình tự DNA
Trên thực tế, giải pháp trên có một vấn đề. Vấn đề là mỗi một lần “đọc”, Sequencer thường chỉ “đọc” được một đoạn ngắn DNA – có độ dài từ 100-1000 bp. Vậy làm thế nào để giải được trình tự của các bộ gen có chiều dài hàng tỷ bp (như gen người có khoảng 3 tỷ bp)?
💡 Có một ý tưởng giải quyết vấn đề này như sau:
- Đầu tiên, người ta nhân bản DNA thành nhiều bản sao y hệt nhau (tham khảo PCR) và cắt các bản sao này thành từng phân mảnh ngắn (cắt một cách ngẫu nhiên) có độ dài chỉ khoảng vài trăm bp
- Các phân mảnh ngắn này cho chạy qua máy Sequencer – máy này “đọc” trình tự DNA của từng phân mảnh, gọi là read
- Sau đó dùng phần mềm để ráp các đoạn đọc được (read) thành chuỗi trình tự gốc ban đầu.
Một cách cô đọng:
DNA → [phân mảnh] → [Sequencer] → [reads] → [ráp các reads] → Trình tự gốc
-
Có thể hình dung chuỗi DNA như một đoạn văn dài. Việc phân mảnh tương tự như việc ta cắt đoạn văn dài đó thành nhiều “phân mảnh” (là đoạn văn ngắn) một cách ngẫu nhiên. Các đoạn văn ngắn không tách rời hoàn toàn mà có phần phủ lên nhau như lợp ngói. Sau đó, căn cứ vào phần phủ lên nhau của các “phân mảnh” (đoạn văn ngắn) chúng ta tìm cách ráp để tìm lại đoạn văn bản gốc ban đầu. Để cho dễ hiểu, tôi xin phép lấy một ví dụ, trích từ tác phẩm “Dế mèn phiêu lưu ký” của nhà văn Tô Hoài. Chúng ta có các “phân mảnh” (đoạn văn ngắn) sau đây:
- một cái hang đất ở bờ ruộng phía bên kia, chỗ trông ra đầm nước
- be đắp tinh tươm thành hang, thành nhà cho chúng tôi từ bao giờ.
- đầm nước mà không biết mẹ đã chịu khó đào bới, be đắp tinh tươm
- Mẹ dẫn chúng tôi đi và mẹ đem đặt mỗi đứa vào một cái hang đất
Phần in đậm là phần phủ lên nhau của các “phân mảnh”. Sau khi ráp các phần phủ lên nhau chúng ta suy ra được văn bản gốc như sau:
Mẹ dẫn chúng tôi đi và mẹ đem đặt mỗi đứa vào một cái hang đất ở bờ ruộng phía bên kia, chỗ trông ra đầm nước mà không biết mẹ đã chịu khó đào bới, be đắp tinh tươm thành hang, thành nhà cho chúng tôi từ bao giờ.
⚠ Chú ý: trên thực tế, ráp trình tự (sequence assembly) là vấn đề rất phức tạp - mời anh/chị tham khảo bài toán “ráp trình tự”.
-
Ý tưởng cắt nhỏ rồi ráp lại như trên có tên gọi là shotgun sequencing (giải trình tự theo phương pháp súng ngắn) được Staden đề xuất lần đầu vào năm 1979 (bài báo). Vào thời điểm của bài báo (1979) và mãi cho đến tận đầu những năm 2000, giới nghiên cứu chỉ biết đến phương pháp giải trình tự của Sanger (Sanger sequencing). Ngay cả dự án khổng lồ Human Genome Project (dự án bản đồ gen người), người ta cũng chỉ biết đến phương pháp của Sanger. Điểm hạn chế cơ bản của phương pháp này là mỗi một lần chạy, máy chỉ đọc được duy nhất một read với chiều dài không quá 1.000 bp. Vì vậy, để có thể đọc được nhiều read, cách duy nhất là phải sử dụng nhiều máy đồng thời, mỗi máy đọc một đoạn DNA khác nhau. Sau đó dùng phần mềm để ráp các read (xem giải thích ở trên).
Mời anh/chị tham khảo các con số thực tế của phương pháp Sanger (nguồn):
- Chiều dài mỗi read: 400-900 bp
- Độ chính xác: 99.9%
- Số lượng read/1 lần chạy: 1
- Thời gian 1 lần chạy: 20 phút – 3 giờ
- Đơn giá để giải trình tự 1 tỷ bp: $2,400,000 (hai triệu bốn trăm nghìn USD)
-
Vì các hạn chế của phương pháp Sanger, các nhà khoa học bắt buộc phải tìm một giải pháp khác với mục tiêu là tăng tốc độ xử lý. Vào khoảng năm 2004-2005, người ta đưa ra một cách tiếp cận mới để giải trình tự DNA có tên là ‘massively parallel’ (xử lý song song quy mô lớn). Dòng công nghệ này được đặt tên là ‘next-generation’ DNA sequencing (Giải trình tự DNA 'thế hệ tiếp theo'), viết tắt NGS. Ý tưởng đột phá của NGS là multiplexing – xử lý đa kênh. Mỗi một lần chạy, phương pháp của Sanger chỉ cho phép 1 phản ứng (reaction) xảy ra trong ống nghiệm. Trong lúc đó, NGS cho phép hàng trăm nghìn, hàng triệu phản ứng xảy ra đồng thời. NGS có thể giải trình tự đồng thời khoảng 500,000 phân mảnh (mỗi phân mảnh có độ dài cỡ hàng trăm bp). Đến thời điểm hiện nay thì NGS đã tiến rất xa so với công nghệ “cổ điển” (phương pháp của Sanger). Dòng công nghệ này có khả năng giải trình tự toàn bộ bộ gen người chỉ trong vòng một ngày! (Nguồn) Vào năm 2019, các hãng dẫn đầu của dòng NGS gồm Illumina, Qiagen và ThermoFisher Scientific, trong đó Illumina chiếm thị phần lớn nhất.
Mời anh/chị tham khảo các con số thực tế của dòng máy Illumina (nguồn):
- Chiều dài mỗi read:
- MiniSeq, NextSeq: 75–300 bp;
- MiSeq: 50–600 bp;
- HiSeq 2500: 50–500 bp;
- HiSeq 3/4000: 50–300 bp;
- HiSeq X: 300 bp
- Độ chính xác: 99.9% (tức là phred30 – tham khảo Phred quality score)
- Số lượng read/1 lần chạy:
- MiniSeq/MiSeq: 1-25 triệu;
- NextSeq: 130-400 triệu;
- HiSeq 2500: 300 triệu - 2 tỷ;
- HiSeq 3/4000: 2.5 tỷ;
- HiSeq X: 3 tỷ
- Thời gian 1 lần chạy: 1-11 ngày
- Đơn giá để giải trình tự 1 tỷ bp: $5 - $150
-
Đối với phương pháp cổ điển (phương pháp Sanger) hoặc thế hệ NGS, khi muốn giải trình tự DNA, chúng ta bắt buộc phải gửi mẫu DNA đến các trung tâm tính toán. Thời gian chờ kết quả phải tính bằng ngày/tuần/tháng. Có cách nào để giải trình tự trực tiếp từ một thiết bị cầm tay không? Các nhà khoa học tiếp tục tìm cách giải trình tự DNA theo cách tiếp cận mới này. Đại diện cho giải trình tự DNA “thế hệ thứ ba” là công nghệ nanopore. Công nghệ này dựa trên ý tưởng ép sợi DNA cho đi qua một lỗ nhỏ trên màng, mỗi một base bật ra giống như một hạt trên một chuỗi. Bằng cách đo sự thay đổi độ dẫn điện khi mỗi base khác nhau di chuyển qua lỗ, từ đó có thể đọc một cách trực tiếp trình tự của sợi DNA. Công nghệ này có thể đọc được các đoạn DNA rất dài lên tới 2,272,580 bp (nguồn).
Mời anh/chị tham khảo các con số thực tế của phương pháp giải trình tự nanopore (nguồn):
- Chiều dài mỗi read: người dùng có thể cài đặt con số này – cao nhất 2,272,580 bp
- Độ chính xác: ~92–97%
- Số lượng read/1 lần chạy: căn cứ theo cài đặt của người dùng
- Thời gian 1 lần chạy: 1 phút – 48 giờ
- Đơn giá để giải trình tự 1 tỷ bp: $7 - $100
🧬
④ De novo sequencing vs. resequencing
De novo dịch từ tiếng Latin có nghĩa là "từ đầu". Khi giải trình tự gen của một loài nào đấy, việc giải trình tự của cá thể đầu tiên trong loài đó được gọi là De novo sequencing – giải trình tự khởi phát từ đầu. Nghĩa là việc giải trình tự không dựa được vào một khung mẫu có sẵn nào cả.
Bây giờ, giả thiết rằng chúng ta đã giải trình tự được một cá thể đầu tiên của một loài (ví dụ đã giải được trình tự DNA của người đầu tiên trong dự án bản đồ gen người). Cá thể tiếp theo cùng loài, rõ ràng là phải có rất nhiều đoạn DNA có trình tự giống với cá thể đầu tiên. Người ta lấy trình tự đã giải được của cá thể đầu tiên làm khung mẫu cho việc giải trình tự các cá thể khác cùng loài. Việc giải trình tự các cá thể khác này được gọi là resequencing.
Lấy ví dụ về dự án HGP (dự án bản đồ gen người). Người đầu tiên cho mẫu DNA là một cá nhân từ thành phố Buffalo, bang New York, Hoa Kỳ có nguồn gốc nửa châu Âu, nửa châu Phi (nguồn). Người đầu tiên resequence bộ gen của mình là Craig Venter vào năm 2007, người tiếp theo là James Watson. (Vào năm 1953, James Watson là đồng tác giả của bài báo khoa học cùng với Francis Crick đề xuất cấu trúc chuỗi xoắn kép của phân tử DNA.)
Cũng xin lưu ý với anh/chị: DNA sequencing là một quá trình tiệm cận. Vì sao vậy? Tất cả các loại thiết bị, quy trình thí nghiệm, phần mềm chưa bao giờ đạt được độ chính xác 100% cả. Lấy ví dụ về các con số thực tế tôi đã trích dẫn ở trên, độ chính xác thuộc loại cao là 99.9%. Với độ chính xác này, khi giải trình tự gen người thì sai số là bao nhiêu bp? Chúng ta dễ dàng tính ra ngay: gen người có khoảng hơn 3 tỷ bp, độ chính xác là 99.9% tương đương với sai số 0.1% = 1/1.000, lấy 3 tỷ nhân với 0.1%: 3.000.000.000 /1.000 = 3.000.000 (ba triệu) bp! Sai số là 3 triệu bp – một sai số đáng kinh ngạc, đúng không ạ!
Sau khi có nhiều cá thể resequencing thì bản đồ gen dần hiện hình rõ nét. Bản đồ gen gồm các đoạn DNA chung cho tất cả các cá thể cùng loài và các đoạn DNA riêng cho từng cá thể. Việc này giống như việc chúng ta vẽ một bức tranh. Nếu chỉ có vài nét thì rất khó hình dung. Càng có nhiều nét vẽ thì bức tranh càng rõ. Số lượng “nhiều nét” của bản đồ gen người phải đạt con số hàng triệu, hàng chục triệu trên ước tính dân số hiện nay của thế giới là khoảng 8 tỷ người.
🧬
⑤ Tương lai của DNA sequencing?
Ngoài loài người ra, trên Trái Đất có bao nhiêu loài sinh vật? Thưa anh/chị tôi có Google và được đáp án khoảng một nghìn tỷ loài (1.000.000.000.000)! Đó là chưa tính các loài đã bị tuyệt chủng. Chú ý rằng hoàn toàn có thể giải trình tự DNA các loài đã bị tuyệt chủng. Xem ra, công việc của các nhà khoa học còn khá nhiều 😊.
Tất nhiên, các nhà khoa học sẽ lấy cái hữu hạn để tiếp cận cái vô hạn. Ví dụ, họ sẽ biến các máy DNA sequencing như là một thiết bị cảm biến (sensor), có thể hiển thị chuỗi DNA trong giây lát, lập phân tích (analysis) và báo cáo tức thì (real-time) lên một App trên thiết bị di động (như chiếc smartphone mà anh/chị đang sở hữu). Tất nhiên, đó là tôi nói về một giả định trong “thì tương lai”. Còn với “thì hiện tại” DNA sequencing được đánh giá chỉ đang ở trong giai đoạn sơ khai.
🧬
⑤++ Nhìn ra ngoài Vũ trụ
Trước khi kết thúc bài post này, tôi xin phép cung cấp anh/chị một thông tin – đó là bài báo với tựa đề “Identifying the wide diversity of extraterrestrial purine and pyrimidine nucleobases in carbonaceous meteorites” đăng trên ‘Nature Communications’ vào ngày 26/4/2022. Đại ý là sau khi phân tích một nhóm thiên thạch, lần đầu tiên các nhà khoa học Nhật Bản tìm thấy năm thành phần cơ bản của DNA trong các thiên thạch này. Nghiên cứu này củng cố giả thuyết rằng nguồn gốc của sự sống trên Trái Đất đến từ không gian ngoài vũ trụ! Vì vậy, việc đọc được mã DNA không những giúp khám phá các bí ẩn của sự sống trên Trái Đất mà có thể giải mã được sự sống (nếu có) trong vũ trụ bao la 😊.
-
Chúc anh/chị đọc vui nhã!
(_/)
( •_•)
/ >☕
-:-
